Pour pouvoir évaluer le plus précisément possible l’ampleur de l’appauvrissement de la biodiversité il faut des indicateurs fiables ; cela implique des progrès techniques importants.
De ce fait, les objectifs de ScaraB'obs sont :
- Élaborer des techniques qui permettent de détecter la présence des espèces à partir des traces que celles-ci laissent dans l’environnement (« ADN environnemental ou ADNe »). Les enjeux porteront principalement sur l’étalonnage de la méthode et la définition de ses limites (fidélité, détectabilité).
- Élaborer des modèles/cartes de distribution potentielle pour chaque espèce pour disposer d’un référentiel nécessaire à l’évaluation de l’état de conservation de la biodiversité. Les modèles seront élaborés à partir des données disponibles dans la base de données nationale. L’état de conservation sera évalué en confrontant la biodiversité potentielle totale (issue des modèles) au résultat des inventaires réalisés grâce à l’ADNe.
- Constituer le réseau de partenaires institutionnels capable de s’approprier les innovations scientifiques et techniques développées pour mettre en œuvre une évaluation dynamique de la biodiversité à l’échelle régionale et nationale (stratégie d’observatoire et de déploiement local/régional).
Vous avez dit ADNe ?
Le monitoring ADNe repose sur la collecte de substrats environnementaux (eau, écorces, sol…) dans lesquels est extrait l’ADN. Celui-ci est ensuite analysé, sur la base du barcoding ou du metabarcoding, pour voir s’il correspond à un ou plusieurs taxons ciblés (Culver et al. 2011 ; Taberlet et al. 2012). Cette méthode repose donc sur l’existence préalable d’une base de référence de « codes barre » (majoritairement fragments d’un gène mitochondrial dit ‘COI’ pour cytochrome oxydase I mais également la séquence d’ARN ribosomique 16S (ARNr 16S)) pour les espèces que l’on souhaite inventorier (Hebert et al. 2003 ; Miller et al. 2016). Cet ADN trouvé dans l’environnement peut provenir d’urine, fèces, sang, salive, poils, plumes, entre autres. Nous pouvons donc parler d’ADN extracellulaire (Miaud et al. 2012 ; Bohmann et al. 2014 ; Thomsen &Willerslev 2015).
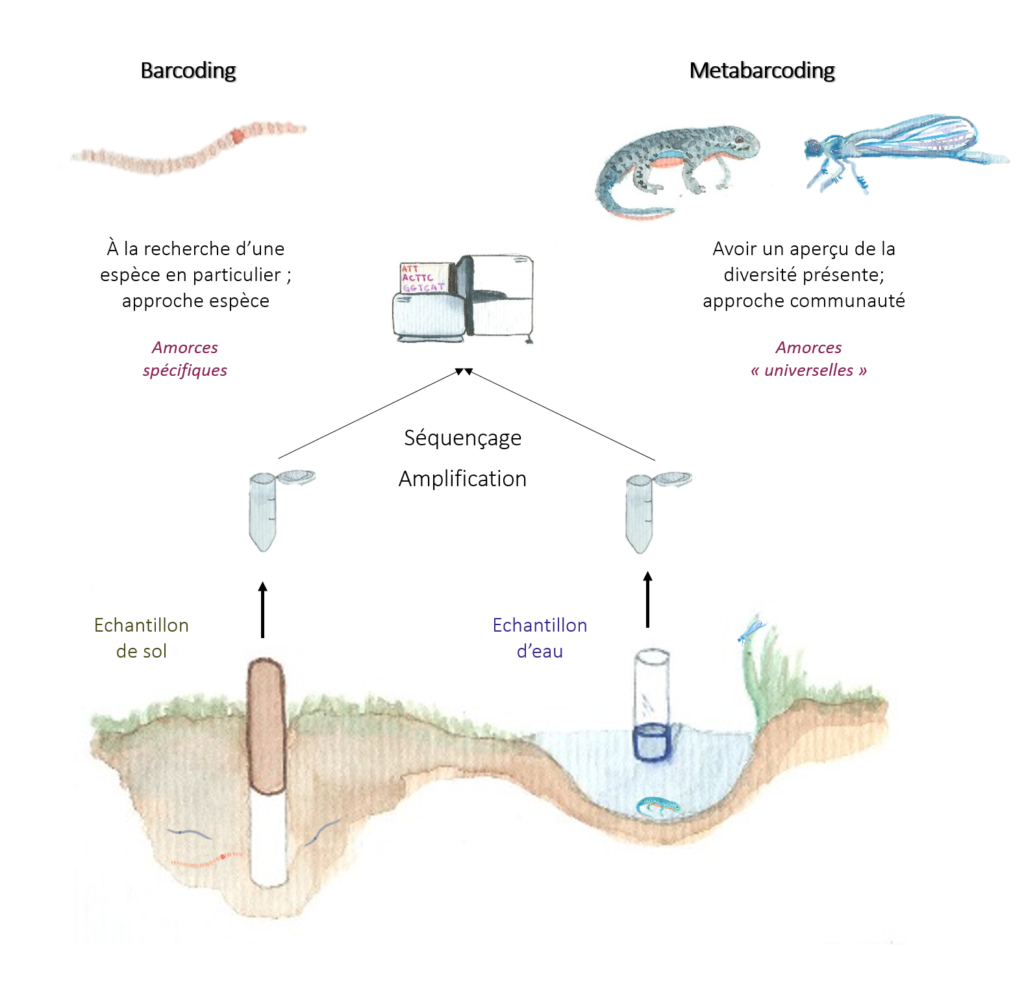
Voici quelques exemples d’organismes dont l’ADN a été collecté dans l’environnement. D’abord il y a prélèvement de l’ADNe dans un milieu ; après l’extraction, l’ADN est amplifié par PCR (Polymérase Chain Reaction), séquencé et enfin confronté aux bases de données informatisées, avant les conclusions sur la détection d’une espèce en particulier (barcoding) ou d’un peuplement dans son ensemble (metabarcoding). Illustration : C. Leandro.
Les inventaires basés sur l’ADNe ont considérablement augmenté à partir de 2010 (Leandro & Miaud 2017) même si la méthode avait déjà été développée dans les années 1980 dans le contexte particulier de l’inventaire de micro-organismes, tels que des bactéries et des champignons (Nanney 1982 ; Hebert et al. 2003 ; Culver et al. 2011; Taberlet et al. 2012). En parallèle, les techniques de séquençage du matériel génétique ont considérablement progressé depuis une vingtaine d’années (Miaud et al. 2012). Il est aujourd’hui possible d’isoler et d’amplifier de très faibles quantités d’ADN extracellulaire plus ou moins dégradé (Hajibabaei et al. 2006 ; Thomsen & Willerslev 2015). De plus, les coûts d’analyse se sont considérablement réduits.
Avec des développements forts et des avantages réels en termes d’automatisation (Gaston & O'Neill 2004), de rapidité et de possibilité d’identification des organismes sans destruction (Hebert & Gregory 2005 ; Lecq et al. 2015), l’ADNe a contribué de façon majeure à la « Révolution Moléculaire » (Lawson Handley 2015) et s’impose comme la méthode de monitoring et détermination de notre siècle (Corlett 2017).
Les suivis basés sur l’ADNe ont été particulièrement développés en milieu aquatique (Ficetola et al. 2008 ; Miaud et al. 2012 ; Spear et al. 2014) ; ces méthodes d’identification se sont largement démocratisées comme un grand nombre de synthèses et guides le démontrent (Miaud et al. 2012 dans Sciences Eaux & Territoires ; Laramie et al. 2015 dans U.S. Geological Survey Techniques and Methods). L’eau est en effet un substrat propice car relativement homogène, notamment dans le cas de systèmes fermés. En effet, la détection de l’ADNe est dépendante de la dispersion des micro-restes portant une information moléculaire dans l’environnement où ils ont été « laissés » (Thomsen et al. 2012 ; Furlan et al. 2015).
Il y a quelques exemples en milieu terrestre (Bienert et al. 2012 ; Thomsen & Willerslev 2015), mais ils restent encore minoritaires, si l’on exclut les inventaires de communautés bactériennes et fongiques dans le sol.
Source : Extrait du Chapitre 2 (partie 2) du manuscrit de thèse de C. LEANDRO (2018) "Conservation de l’entomofaune ordinaire : enjeux scientifiques et sociétaux".

